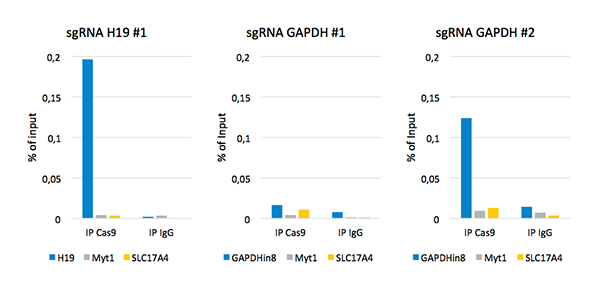
ChIP was performed on sheared chromatin from 4,000,000 HEK293T cells using the iDeal ChIP-seq Kit for Transcription Factors, 5 µl of the polyclonal Cas9 antibody and 1 µg of the negative control IgG. Primers specific for the human H19, GAPDH, Myt1 and SLC17A4 were used for the qPCR. The figure shows the recovery, expressed as a percent of input (the relative amount of immunoprecipitated DNA compared to input after qPCR analysis).
ゲノム編集CRISPR/Cas9
ChIPによるガイドRNAの選択
様々なツールが特定の遺伝子座をターゲットとするsgRNAの研究に役立ちますが、Cas9と目的のsgRNAがゲノムに特異的に結合するかを正確に予測することが課題とされています。
CRISPR / Cas9を特定したターゲティングを確実にするために、目的の遺伝子座でのsgRNAの特異的結合の検証が必要です。 クロマチン免疫沈降とそれに続くリアルタイムPCR(ChIP-qPCR)またはシーケンシング(ChIP-seq)は、このような検証に最適なでありタンパク質とDNAの相互作用を解析します。
DiagenodeのiDealChIP-seq Kit for Transcription Factorsは、ChIPとその後に行うリアルタイムPCRによってオンターゲットな領域のdCas9を特異的に濃縮することで、ゲノム内のdCas9結合を調査する堅牢なワークフローが特徴です。さらに、9dCas抗体で免疫沈降したDNAのシーケンス解析は、エフェクターに融合したdCas9の正しい結合を確認するために不可欠であるゲノムワイドなオフターゲット効果の可能性を示しています。
1. Monitoring of sgRNA efficiency by ChIP-qPCR
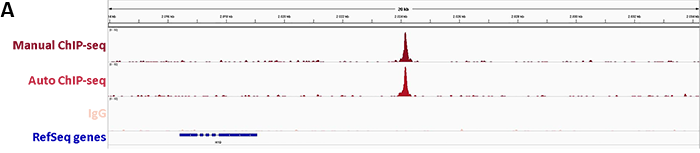
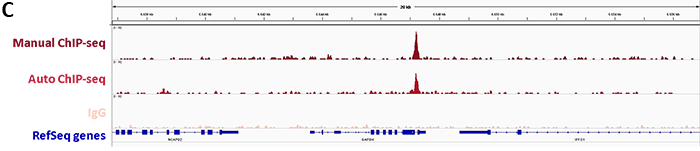
2. Confirmation of binding specificity of sgRNA and detection of off-target bindings by ChIP-seq